-
데이터 변경이력
데이터 변경이력 버전 일자 변경내용 비고 1.2 2023-11-15 데이터 최종 개방 1.1 2023-06-07 非안심존 데이터 전환 1.0 2023-04-30 데이터 개방(Beta Version) 데이터 히스토리
데이터 히스토리 일자 변경내용 비고 2025-05-08 구축업체정보 수정 2023-12-08 산출물 전체 공개 소개
더 빠르고 안전한 신약개발을 위해 바이오·의료 논문 기반 생물학적 상호 연관성 데이터 추출 및 분석 인공지능 신약개발 솔루션(Bio NLP) 개발을 위한 학습 데이터 구축 사업
구축목적
2만개의 논문 내 생물학적 상호 연관성 정보를 추출하여 Bio NLP 인공지능 학습 데이터를 구축함. (5대 질환별 4천 개, 총 2만 개의 데이터 구축)
-
메타데이터 구조표 데이터 영역 한국어 데이터 유형 텍스트 데이터 형식 pdf 데이터 출처 PubMed 라벨링 유형 색인, 주석(텍스트) 라벨링 형식 JSON 데이터 활용 서비스 생물학적 상호 연관성 분석, Pathway, Bio NLP, 바이오·의료 논문 분석 데이터 구축년도/
데이터 구축량2022년/20,000건 -
데이터 통계
데이터 구축 규모
데이터 구축 규모 데이터 종류 원문 데이터 원문 데이터 규모 가공 데이터 규모 최종 데이터 구축 규모 형태 대장암 PDF 4,000 건 7,600 건 4,000 건 (Colorectal cancer) 유방암 PDF 4,000 건 7,600 건 4,000 건 (Breast cancer) 당뇨병 PDF 4,000 건 7,600 건 4,000 건 (Diabets) 전립선암 PDF 4,000 건 7,600 건 4,000 건 (Prostatic carcinoma) 심근경색 PDF 4,000 건 7,600 건 4,000 건 (MI) 총계 20,000 건 38,000 건 20,000 건 데이터 분포
* 구축 데이터 용도별 데이터 분포
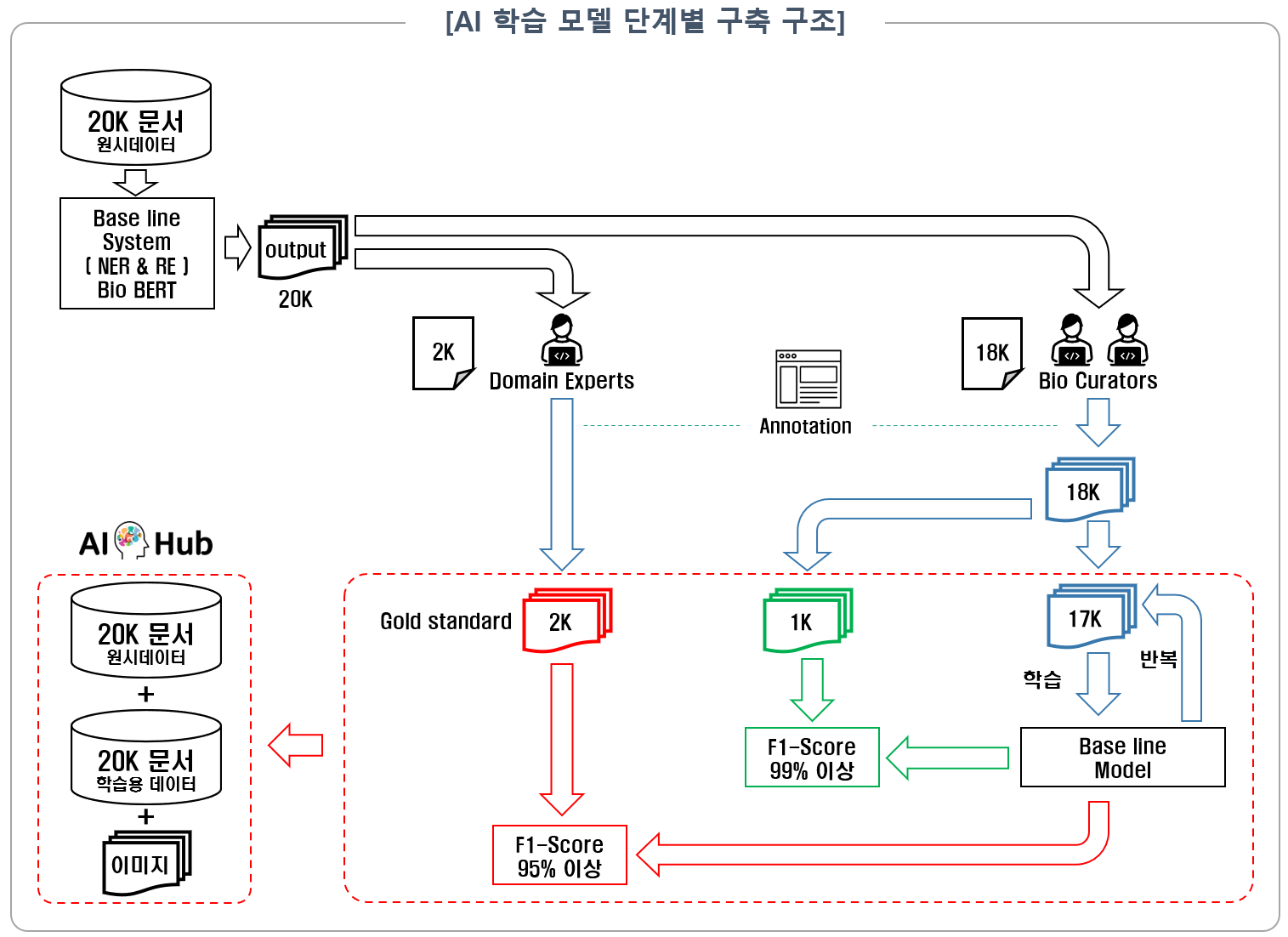
데이터 분포 구분 포맷 수량 비율 AI 학습모델 데이터 분포 학습용 데이터 JSON 18,000 건 90% 테스트 데이터 JSON 2,000 건 10% 합계 20,000 건 100% 모델 학습 과정별 데이터 분포 Training Set JSON 17,000 건 85% Validation Set JSON 1,000 건 5% Test Set JSON 2,000 건 10% 합계 20,000 건 100% * 학습용 데이터(18,000개)와 테스트 데이터(2,000개)의 관계 정보 비율
학습용 데이터 관계명 학습 데이터 비율 테스트 데이터 비율 Associate 19,788 36.43% 2,496 38.22% Reduces 3,637 6.70% 446 6.83% Suppress 2,752 5.07% 301 4.61% Inhibit 9,038 16.64% 992 15.19% Regulate 7,476 13.76% 838 12.83% Stimulate 5,801 10.68% 679 10.40% Treat 2,485 4.58% 306 4.69% Prevent 1,494 2.75% 208 3.19% Biomarker 1,845 3.40% 264 4.04% Total 54,316 100.00% 6,530 100.00% -
-
AI 모델 상세 설명서 다운로드
AI 모델 상세 설명서 다운로드 AI 모델 다운로드활용 모델
모델학습
인공지능 기반 바이오·의료 논문 간 상호연관성 추출 모델의 데이터 구축 구성도는 아래 그림과 같음. 모델 학습 과정별 데이터는 학습용(Training) 85%, 검증용(Validation) 5%, 시험용(Test) 10% 의 비율로 구성함.
서비스 활용 시나리오
∎ General Bio-NLP 개발
본 과제의 학습데이터, 모델을 바탕으로 바이오·의료 논문 기반 자연어처리(NLP) 인공지능 서비스를 개발할 수 있음. 논문을 비롯한 비정형 데이터를 분석하고 상호연관성을 추출하여 신약개발 및 의료 연구에 사용될 수 있음
∎ 질병 특화 NLP(NER/RE) 개발
본 과제에서 구축한 5대 질병 중 하나를 선택하여 확장하거나 동일 구조로 다른 질병의 데이터를 학습시켜서 모델을 개발할 수 있으며 그러한 모델은 특정 질병에 특화된 세분화되고 자세한 예측 결과를 도출 할 수 있음.
∎ Knowledge Graph based AI Drug Discovery 개발
신약개발 인공지능 솔루션 개발 업체에서 Knowledge Graph 기반의 Drug Discovery 인공지능 솔루션 및 서비스를 개발함에 있어서 본 과제의 학습 데이터 및 모델이 활용될 수 있음.기타 정보
유의사항
∎ 저작권 관련
논문 데이터를 학습 데이터로 사용함에 있어 각 논문별로 선언하는 저작권 방식을 구체적으로 확인하여 적용해야 함. ‘수집’, ‘수정’, ‘재배포’, ‘상업성 유무’ 등의 라이선스 조건에 맞춰 저작권 관리가 필요하며, 사전 전문 법무법인 혹은 변호사 법률 검토를 권장함.
∎ 개인정보 처리
논문을 비롯한 비정형 데이터를 수집, 가공하는 과정에서 논문의 저자 이름 등의 개인 정보는 저작권과 다른 법적 검토를 통해 사용해야 함. 가능한 사용하지 않는 것을 권장함.
∎ 학습데이터 품질관리
논문 데이터를 학습 데이터로 사용할 때 가능한 권위가 있는 소스를 통해서 원시데이터를 수집해야 함. 학습모델의 성격상 전혀 다른 결과를 도출하는 신뢰할 수 없는 학습데이터의 양이 많아질수록 전체 네트워크의 품질이 현저하게 떨어짐. -
데이터 성능 점수
측정값 (%)기준값 (%)데이터 성능 지표
데이터 성능 지표 번호 측정항목 AI TASK 학습모델 지표명 기준값 점수 측정값 점수 1 바이오, 의료 분야 개체명 인식 기반 개체 간 관계추출 모델 성능 Prediction BERT F1-Score 0.95 점 0.9638 점
※ 데이터 성능 지표가 여러 개일 경우 각 항목을 클릭하면 해당 지표의 값이 그래프에 표기됩니다.
※ AI모델 평가 지표에 따라 측정값의 범위, 판단 기준이 달라질 수 있습니다. (ex. 오류율의 경우, 낮을수록 좋은 성능을 내는 것으로 평가됩니다)
-
설명서 및 활용가이드 다운로드
데이터 설명서 다운로드 구축활용가이드 다운로드데이터 포맷
원문데이터 포맷 예시
원문데이터 포맷 예시 제목 CYP17 5'-UTR MspA1 polymorphism and the risk of premenopausal breast cancer in a German population-based case–control study 논문 고유번호 PMC1175058 카테고리 Breast cancer(유방암) 논문 발행기관 BioMed Central Ltd. 논문 학술지 Breast Cancer Research 논문 참조 링크 https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1175058/ 원문(PDF) 
속성 및 내용 구분 No 속성명 속성 및 내용 필수 1 sourcid 해당 문서의 PMC 고유번호 필수 2 link 해당 문서 원본 URL 필수 3 text 문서에서 추출된 Text 필수 4 entities[] 문서의 모든 Entity 정보 필수 5 entityId Entity ID로서 Entity의 시작글자 자릿수_끝글자 자릿수 필수 6 span Entity의 위치 정보 필수 7 begin Entity의 시작글자 자릿수 필수 8 end Entity의 끝글자 자릿수 필수 9 entityType Entity의 분류 정보 필수 10 entityName Entity에 해당되는 텍스트 필수 11 id Entity 레퍼런스 정보 필수 12 relation_info[] Relation 정보 필수 13 subjectID Subject에 해당되는 Entity ID 필수 14 subjectText Subject에 해당되는 Text 필수 15 rel_name Subject와 Object의 관계 분류 (타입) 필수 16 object_ID Object에 해당되는 Entity ID 필수 17 objectText Object에 해당되는 Text 선택 1 publisher 논문(해당 문서) 발행기관 정보 선택 2 journal 논문(해당 문서) 발표 학술지 정보 어노테이션 포맷
어노테이션 포맷 구분 속성명 타입 필수 설명 여부 1 sourcid string Y 원시데이터 고유번호 2 publisher string 논문 발행기관 3 journal string 학술지 4 link string Y 원시데이터 참조 링크 5 text string Y 원시데이터 텍스트 추출 내용 6 entities[] Y Entity 정보 6-1 entityId string Y Entity별 고유아이디 6-2 span object Y Entity 위치 정보 6-2-1 begin number Y 텍스트의 시작 위치 6-2-2 end number Y 텍스트의 종료 위치 6-3 entityType string Y Entity 분류 정보 6-4 entityName string Y Entity에 해당되는 텍스트 6-5 id string Y Entity 레퍼런스정보 7 relation_info[] Y Relation 정보 7-1 subjectID string Y Arg1에 해당되는 EntityID 7-2 subjectText string Y Arg1에 해당되는 텍스트 7-3 rel_name string Y 관계 분류 7-4 objectID string Y Arg2에 해당되는 EntityID 7-5 objectText string Y Arg2에 해당되는 텍스트 실제 예시
실제 예시 JSON 형식
{ "sourcid": "Breast cancer(유방암)",
"publisher": "BioMed Central",
"journal": "Breast Cancer Research",
"link": "https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1175058/",
"text": "CYP17 5'-UTR MspA1 polymorphism and the risk of premenopausal breast cancer in a German population-based case-control study Introduction Studies on the association between the cytochrome P450c17alpha gene (CYP17) 5'-untranslated region MspA1 genetic polymorphism and breast cancer risk have yielded inconsistent results. Higher levels of estrogen have been reported among young nulliparous women with the A2 allele. Therefore we assessed the impact of CYP17 genotypes on the risk of premenopausal breast cancer, with emphasis on parity. Methods We used data from a population-based case-control study of women aged below 51 years conducted from 1992 to 1995 in Germany. Analyses were restricted to clearly premenopausal women with complete information on CYP17 and encompassed 527 case subjects and 904 controls, 99.5% of whom were of European descent.
[원천데이터의 텍스트 내용이 이어짐, 이하 생략]
(Entity 시작)
"entities": [
{
"entityId": "0_12",
"span": {
"begin": 0,
"end": 12
},
"entityType": "gene",
"entityName": "CYP17 5'-UTR",
"id": "UMLS:C1420099"
},
{
"entityId": "48_75",
"span": {
"begin": 48,
"end": 75
},
"entityType": "disease",
"entityName": "premenopausal breast cancer",
"id": "UMLS:C0741682"
},
[원천데이터 전체의 Entity가 이어짐, 이하 생략]
(Relation 시작)
"relation_info": [
{
"subjectID": "1080_1091",
"subjectText": "CYP17 A1/A2",
"rel_name": "associate",
"objectId": "1170_1183",
"objectText": "breast cancer"
},
{
"subjectID": "1461_1463",
"subjectText": "A1",
"rel_name": "associate",
"objectId": "1537_1547",
"objectText": "A2 alleles"
}
]
}예시 화면 이미지 
-
데이터셋 구축 담당자
수행기관(주관) : (주)아리바이오
수행기관(주관) 책임자명 전화번호 대표이메일 담당업무 김진우 02-2637-0009 / 070-4128-4402 zinukim@aribio.com 총괄책임자 수행기관(참여)
수행기관(참여) 기관명 담당업무 ㈜로뎀마이크로시스템 작업 플랫폼 개발, 데이터 수집 및 기타 S/W개발 데이터 관련 문의처
데이터 관련 문의처 담당자명 전화번호 이메일 이준원 02-2637-0009 / 070-4128-4402 jwlee@aribio.com
-
인터넷과 물리적으로 분리된 온라인·오프라인 공간으로 의료 데이터를 포함하여 보안 조치가 요구되는 데이터를 다운로드 없이 접근하고 분석 가능
* 온라인 안심존 : 보안이 보장된 온라인 네트워크를 통해 집, 연구실, 사무실 등 어디서나 접속하여 데이터에 접근하고 분석
* 오프라인 안심존 : 추가적인 보안이 필요한 데이터를 대상으로 지정된 물리적 공간에서만 접속하여 데이터에 접근하고 분석 -
- AI 허브 접속
신청자 - 안심존
사용신청신청자신청서류 제출* - 심사구축기관
- 승인구축기관
- 데이터 분석 활용신청자
- 분석모델반출신청자
- AI 허브 접속
-
1. 기관생명윤리위원회(IRB) 심의 결과 통지서 [IRB 알아보기] [공용IRB 심의신청 가이드라인]
2. 기관생명윤리위원회(IRB) 승인된 연구계획서
3. 신청자 소속 증빙 서류 (재직증명서, 재학증명서, 근로계약서 등 택1)
4. 안심존 이용 신청서 [다운로드]
5. 보안서약서 [다운로드]
※ 상기 신청서 및 첨부 서류를 완비한 후 신청을 진행하셔야 정상적으로 절차가 이루어집니다. -
신청 및 이용관련 문의는 safezone1@aihub.kr 또는 02-525-7708, 7709로 문의
데이터셋 다운로드 승인이 완료 된 후 API 다운로드 서비스를 이용하실 수 있습니다.
API 다운로드 파일은 분할 압축되어 다운로드 됩니다. 분할 압축 해제를 위해서는 분할 압축 파일들의 병합이 필요하며 리눅스 명령어 사용이 필요합니다.
리눅스 OS 계열에서 다운로드 받으시길 권장하며 윈도우에서 파일 다운로드 시 wsl(리눅스용 윈도우 하위 시스템) 설치가 필요합니다.
※ 파일 병합 리눅스 명령어
find "폴더경로" -name "파일명.zip.part*" -print0 | sort -zt'.' -k2V | xargs -0 cat > "파일명.zip"
- 해당 명령어 실행 시, 실행 경로 위치에 병합 압축 파일이 생성됩니다.
- 병합된 파일 용량이 0일 경우, 제대로 병합이 되지 않은 상태이니 "폴더경로"가 제대로 입력되었는지 확인 부탁드립니다.
※ 데이터셋 소개 페이지에서 다운로드 버튼 클릭하여 승인이 필요합니다.
오프라인 데이터 이용 안내
본 데이터는 K-ICT 빅데이터센터에서도 이용하실 수 있습니다.
다양한 데이터(미개방 데이터 포함)를 분석할 수 있는 오프라인 분석공간을 제공하고 있습니다.
데이터 안심구역 이용절차 및 신청은 K-ICT빅데이터센터 홈페이지를 참고하시기 바랍니다.

국방데이터 개방 안내
본 데이터는 국방데이터로 군사 보안에 따라 AI허브에서 데이터를 제공하지 않으며,
군 담당자를 통한 별도의 사용 신청이 필요합니다.